????欢迎来到本博客❤️❤️??
?博主优势:???博客内容尽量做到思维缜密,逻辑清晰,为了方便读者。
⛳️座右铭:行百里者,半于九十。
???本文目录如下:???
目录
?1 概述
?2 运行结果
?3 参考文献
?4 Matlab代码、数据、文献
?1 概述
由于各种肿瘤类型,在磁共振图像(MRI)中准确分割脑肿瘤是一项艰巨的任务。使用来自多模态MRI的信息和特征,包括结构MRI和来自扩散张量成像(DTI)的各向同性(p)和各向异性(q)分量,可以对大脑图像进行更准确的分析。方法:我们提出了一种新的基于3D超体素的学习方法,用于分割多模态MRI脑图像(常规MRI和DTI)中的肿瘤。超体素是使用多模态 MRI 数据集中的信息生成的。对于每个超体素,提取各种特征,包括文本描述符的直方图,使用一组具有不同大小和方向的Gabor滤波器计算,以及一阶强度统计特征。这些特征被输入到随机森林(RF)分类器中,将每个超体素分类为肿瘤核心,水肿或健康脑组织。结果:该方法在两个数据集上进行评估:1)我们的临床数据集:11张患者的多模态图像和2)BRATS 2013临床数据集:30张多模态图像。对于我们的临床数据集,使用多模态MRI检测肿瘤(包括肿瘤核心和水肿)的平均灵敏度为86%,平衡错误率(BER)为7%;而自动肿瘤分割与地面真相的骰子评分为 0.84。BRATS 2013数据集的相应结果分别为96%、2%和0.89。结论:该方法在脑肿瘤的分割中显示出有希望的结果。从多模态MRI图像中添加特征可以大大提高分割精度。该方法与所有肿瘤等级的专家描述非常匹配,从而提供了一种更快、更可重复的脑肿瘤检测和描述方法,以帮助患者管理。
?2 运行结果
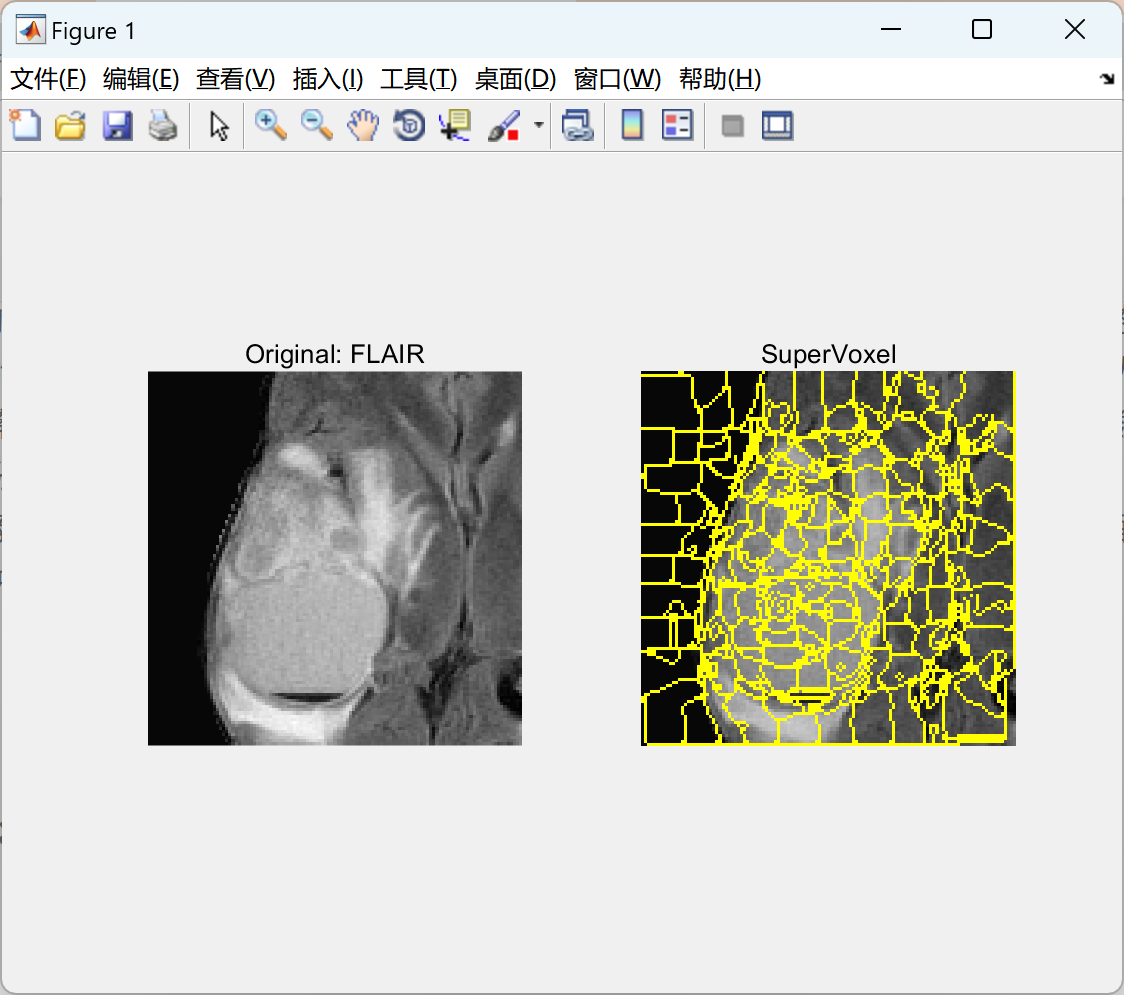


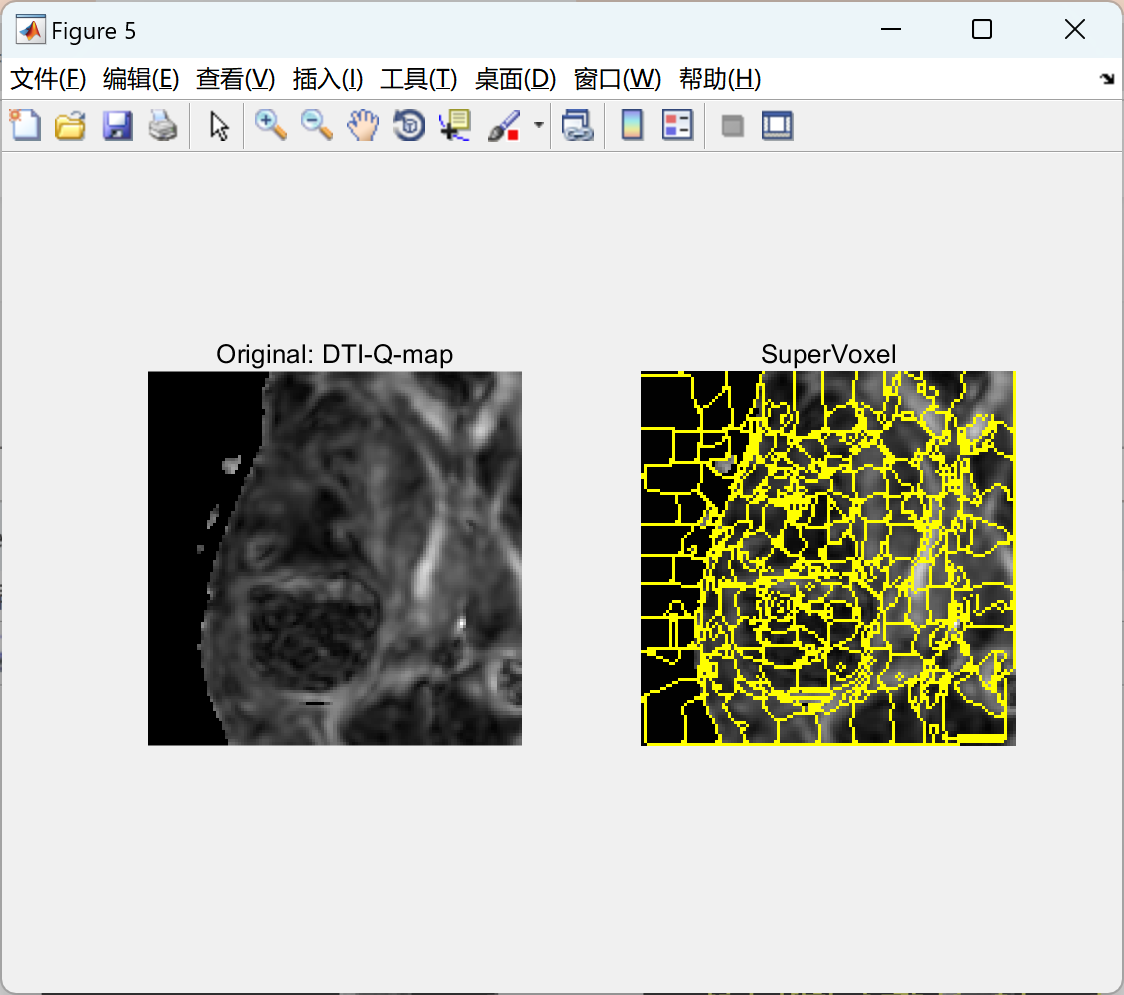
部分代码:
%% Save
% Save the supervoxel map volumes into MAT file
Output_Name = fullfile(Output_Path,['MRI_SLIC_Labels_Size',num2str(voxel_X),...
'x',num2str(voxel_Y),'x',num2str(voxel_Z),'_Compactness_0',Cmpt,'_Case_',num2str(Case),'.mat']);
save (Output_Name,'SLIC_Labels_3D');
%% Show the output
Slice = round(size(I,3)/2);
Image_2D = I(:,:,Slice,1);
Label1 = Label(:,:,Slice,1);
k1 = unique(Label1);
Label2 = zeros(size(Image_2D));
BW = zeros(size(Image_2D));
BW = logical(BW);
for idx = 1:numel(k1) % 1:k
c_k = k1(idx);
L = zeros(size(Image_2D));
L(Label1==c_k)=1;
BW2 = L;
BW_temp = edge(BW2);
Label2 = Label2+double(BW2)*c_k;
BW = BW|BW_temp;
end
for P = 1:numel(ProtocolList)
Image_2D = I(:,:,Slice,P);
BW_Color = repmat(Image_2D,1,1,3);
BW_Color = uint8(BW_Color*255);
for layer = 1:2
tempLayer = BW_Color(:,:,layer);
tempLayer(BW) = 255;
BW_Color(:,:,layer) = tempLayer;
end
tempLayer = BW_Color(:,:,3);
tempLayer(BW) = 0;
BW_Color(:,:,3) = tempLayer;
figure(P);
subplot(1,2,1); imshow(Image_2D,[])
title(['Original: ',ProtocolList{P}])
subplot(1,2,2); imshow(BW_Color,[])
title('SuperVoxel')
end
%% Save
% Save the supervoxel map volumes into MAT file
Output_Name = fullfile(Output_Path,['MRI_SLIC_Labels_Size',num2str(voxel_X),...
'x',num2str(voxel_Y),'x',num2str(voxel_Z),'_Compactness_0',Cmpt,'_Case_',num2str(Case),'.mat']);
save (Output_Name,'SLIC_Labels_3D');
%% Show the output
Slice = round(size(I,3)/2);
Image_2D = I(:,:,Slice,1);
Label1 = Label(:,:,Slice,1);
k1 = unique(Label1);
Label2 = zeros(size(Image_2D));
BW = zeros(size(Image_2D));
BW = logical(BW);
for idx = 1:numel(k1) % 1:k
c_k = k1(idx);
L = zeros(size(Image_2D));
L(Label1==c_k)=1;
BW2 = L;
BW_temp = edge(BW2);
Label2 = Label2+double(BW2)*c_k;
BW = BW|BW_temp;
end
for P = 1:numel(ProtocolList)
Image_2D = I(:,:,Slice,P);
BW_Color = repmat(Image_2D,1,1,3);
BW_Color = uint8(BW_Color*255);
for layer = 1:2
tempLayer = BW_Color(:,:,layer);
tempLayer(BW) = 255;
BW_Color(:,:,layer) = tempLayer;
end
tempLayer = BW_Color(:,:,3);
tempLayer(BW) = 0;
BW_Color(:,:,3) = tempLayer;
figure(P);
subplot(1,2,1); imshow(Image_2D,[])
title(['Original: ',ProtocolList{P}])
subplot(1,2,2); imshow(BW_Color,[])
title('SuperVoxel')
end
?3 参考文献
文章中一些内容引自网络,会注明出处或引用为参考文献,难免有未尽之处,如有不妥,请随时联系删除。文章来源:https://uudwc.com/A/20jnZ
 文章来源地址https://uudwc.com/A/20jnZ
文章来源地址https://uudwc.com/A/20jnZ

