? 课程:生物信息学_玉泉路_课堂笔记 中科院_2022秋季课 第一学期
? 个人笔记使用
? 2023/7/3
? 教材使用:

文章来源地址https://uudwc.com/A/DzZEn
生物信息学是应用计算机科学和数学方法研究生物学问题的交叉学科。它包括了多个领域,如序列分析、结构生物学、基因表达、代谢通路、系统生物学等,涉及到DNA、RNA、蛋白质等生物分子的结构、功能、演化和组织等方面。
? 生物信息学的应用非常广泛,其中包括:
基因组学研究:生物信息学工具可以帮助研究人员在大规模的基因组中进行快速、高效地基因识别和注释,有助于深入了解基因组结构和功能。
蛋白质结构预测:生物信息学可以预测蛋白质的结构,提供给研究人员进一步进行分子模拟和药物研发的基础。
基因表达分析:通过生物信息学分析基因表达数据,可以了解生物体对不同环境和刺激的反应,为疾病的诊断和治疗提供理论基础。
药物研发:生物信息学可以加速药物研发过程,例如通过分析蛋白结构,寻找靶点并提供可行的药物设计方案。
最新的发展方向包括实时数据处理、人工智能、深度学习、量子计算等。生物信息学研究领域的数据量不断增加,在快速处理和分析大型数据方面,实时数据处理技术有助于提高数据处理和分析的效率。
另外,人工智能和深度学习技术可以更好地处理现实的不确定性和复杂性,推动整个生物信息学领域的发展。
1 生物信息学早期应用
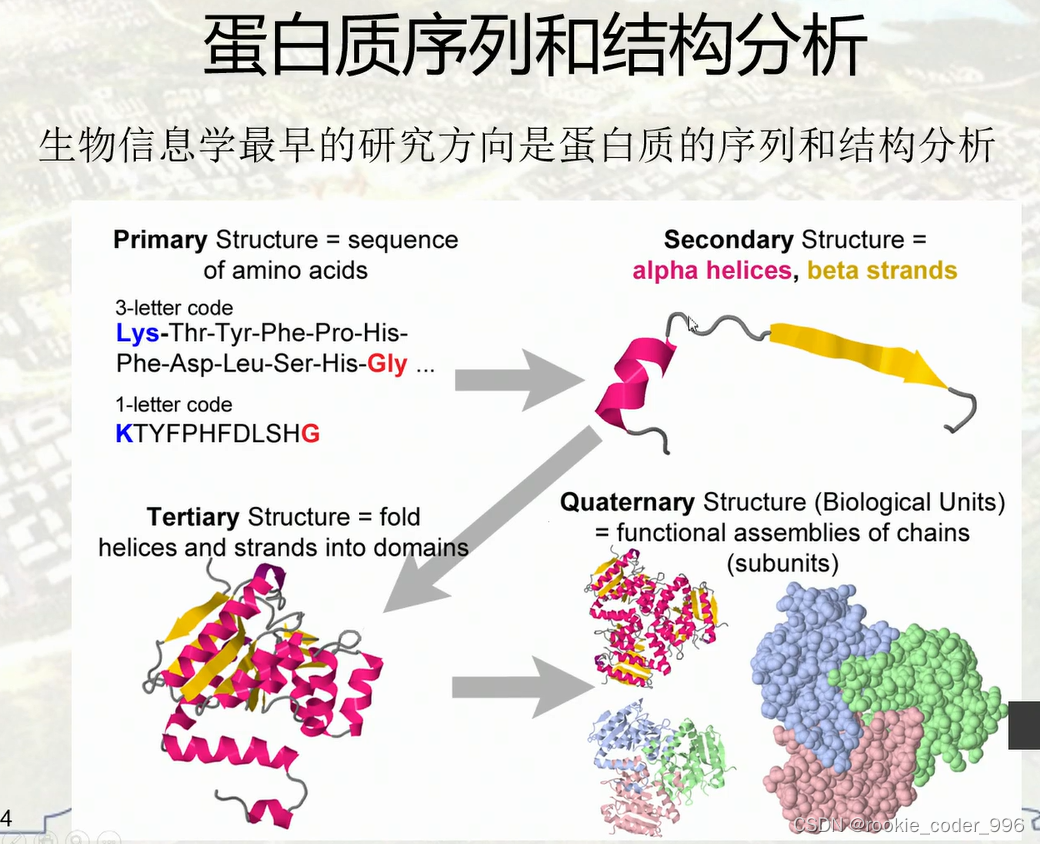
蛋白质序列和结构分析是研究蛋白质分子结构的领域。蛋白质是生命体中最基本的功能分子之一,其中的氨基酸序列和三维结构对于其功能至关重要。
蛋白质序列分析是研究蛋白质序列的特征和性质,以及推断其功能和演化。常用的方法包括比对、聚类、模拟、? 机器学习等。
蛋白质结构分析是研究蛋白质的三维结构,包括其形态、构象和动力学特性。常用的方法包括X光晶体学、核磁共振、电子显微镜、质谱等。
蛋白质序列和结构分析的应用非常广泛,涵盖了生物医学、食品科学、生物工程等多个领域。例如,它们可以用于药物研究、基因工程、食品质量控制等。
1951 年,Linus Pauling (莱纳斯·鲍林)和 Robert Corey(罗伯特·科里) 提出了 α-螺旋和 β-折叠是蛋白质二级结构的基本构建单元的理论。
1955年,英国生物化学家Frederick·Sanger (弗雷德里克·桑格)将胰岛素的氨基酸序列完整测定出来,得到了 51个氨基酸的牛胰岛素蛋白序列,为此他于 1958 年获得了诺贝尔化学奖。
1958年,当英国科学家 John Kendrew 和 Max Perutz(约翰·肯德鲁和马克斯·佩鲁兹) 发表了第一个高分辨率蛋白质结构,首先是肌红蛋白,然后是血红蛋白
1962年,Linus Pauling (鲍林)提出基于蛋白质序列的分子进化理论
? 标志着生物信息学的来临。
1965年,Margaret Dayhof (玛格丽特戴霍夫)构建了第一个分子数据库:蛋白质序列数据库。
2 序列匹配算法

序列匹配算法最早用在蛋白质氨基酸序列的比较,后来扩展到了核酸序列的比较
生物信息学的序列匹配算法是用于比较和发现DNA、RNA或蛋白质序列之间的相似性和匹配关系的算法。这些算法可以用来识别相似的或相关的序列,从而推断它们之间的功能或进化关系。
最常见的生物信息学序列匹配算法包括:
?1. 基于相似性的搜索算法(例如,BLAST):这些算法通过比较查询序列与数据库中已知序列的局部相似性来查找最佳匹配。它们使用快速的启发式方法来提高搜索效率,通常用于快速发现高度相似的序列。
?2. 全局序列比对算法(例如,Needleman-Wunsch算法):这些算法通过比较两个序列的所有可能组合来实现全局匹配。它们根据序列中的相似性和差异性建立得分矩阵,然后使用动态规划算法来识别最佳的序列匹配。
?3. 局部序列比对算法(例如,Smith-Waterman算法):这些算法仅考虑序列中的局部相似性,并对相似片段进行比对和得分。它们在查找较长序列中的局部匹配时非常有用。
?4. 多序列比对算法(例如,ClustalW):这些算法用于比较和对齐多个序列。它们可以帮助识别序列中的保守区域和变异位点,以及推断序列之间的进化关系。
这些序列匹配算法在生物信息学的各个领域中被广泛使用,包括基因组学、蛋白质组学和分子进化研究。它们帮助研究人员理解生物分子的结构、功能和进化关系,以及推断蛋白质的结构和功能。
3 PDB数据库
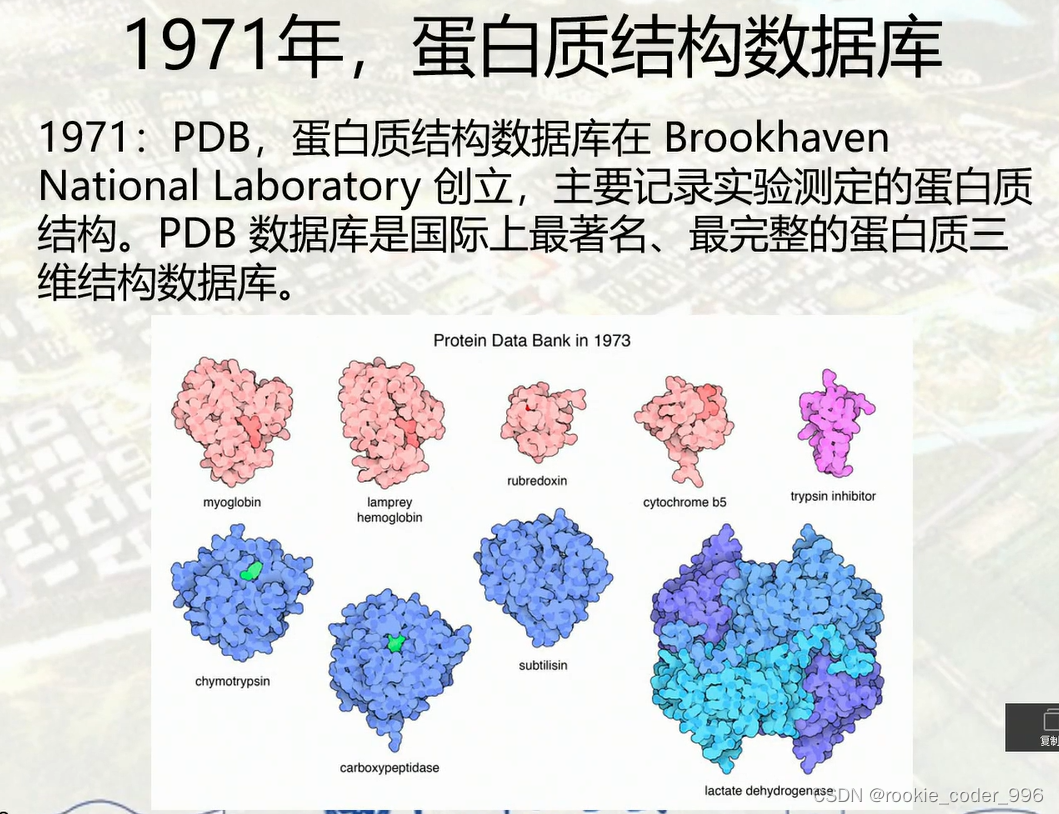

PDB(Protein Data Bank)是一个专门用于储存生物大分子三维结构数据的数据库,包括蛋白质、核酸以及其他生物大分子的结构信息,目前是全球最大的生物大分子三维结构数据库之一。
PDB数据库的数据来源主要来自于X射线晶体学、NMR(核磁共振)以及其他方法获取的生物大分子三维结构数据。
这些数据通过专业的算法和协议进行整理和管理,形成了统一的文件格式,即PDB文件。PDB文件中包括了生物大分子原子坐标、拓扑结构信息等重要信息,同时也含有相关的文献引用和其他元数据。
PDB数据库的应用十分广泛,包括了生物学、化学、药物研究等领域。在生物学研究中,PDB数据库可以帮助科学家更好地理解蛋白质和其他生物大分子的结构与功能之间的关系,从而加深对生物学机理的认识。
在药物研究中,PDB数据库可以用来帮助寻找药物的作用靶点以及设计更好的药物分子。
4 序列比对算法

BLAST算法,是测定一个序列之后,可以用来和数据库当中所有的序列进行比对的算法,在核酸序列比对中常用,当然最初的时候是对于氨基酸序列的比较,最后扩展使用到核酸序列上面
BLAST (Basic Local Alignment Search Tool) 算法是一种常用的序列比对算法,用于比较两个或多个生物序列的相似性,以识别最相似的序列或确定序列间的进化关系。
BLAST算法是一种快速、高效的序列比对方法,广泛应用于生物信息学、基因组学、蛋白质学等领域。
BLAST算法的基本思想是将查询序列与参考序列库中的所有序列进行比对,寻找最相似的序列,从而确定两个序列的相似性。
BLAST算法的主要步骤包括:
1. 预处理:将参考序列库中的所有序列进行处理,并生成索引文件以提高搜索速度。
2. 查询序列比对:将查询序列拆分成多个较小的子序列,与参考序列库中的序列进行比对。
3. 比对结果评估:根据比对结果,计算两个序列间的相似性得分。
4. 结果输出:将比对结果按照得分高低排序并输出。
? BLAST算法采用了一种局部比对的策略,即先找到最好的匹配子序列,再进行全局比对,从而节省时间和计算资源。此外,BLAST算法还采用了一种启发式的搜索策略,通过设置一些限制条件,限制搜索范围,减少搜索时间。这些限制条件包括:序列长度、匹配长度、失配长度和间隙长度等。
5 蛋白质结构域数据库 和 CASP比赛

Motif 是指一小段连续的氨基酸或核苷酸序列,它是构成功能域的功能单元
若干个 Motif 组成一个域 domain

6 蛋白质组学

蛋白质组学是研究生物体内所有蛋白质的组成、结构、功能、相互作用及调控的一门学科。蛋白质是生物体内最基本的分子机器,扮演着重要的生物学功能,而蛋白质组学的主要目标是全面了解蛋白质在生物系统中的作用和调节机制,以及其在疾病发生和治疗中的作用。
蛋白质组学的研究背景是因为人们意识到基因组学研究的局限性,即同样的基因可以转录成不同的蛋白质,不同的蛋白质可能发挥不同的功能。因此,研究蛋白质组成和功能有利于深入了解生物体各个层次的生命现象。
蛋白质组学的研究方向主要有以下几个方面:
1. 蛋白质组成分析:通过高通量技术,全面分析生物体内的蛋白质组成及其表达量等信息,以揭示细胞、组织、器官和生物体的蛋白质表达特征。
2. 蛋白质结构研究:通过结构生物学技术,研究蛋白质三维结构及其与其他分子之间的相互作用,以理解蛋白质的功能和调控机制。
3. 蛋白质交互作用研究:通过蛋白质组学技术,研究蛋白质与蛋白质、蛋白质与其他分子之间的相互作用网络,以揭示生命分子之间的相互作用网络。
4. 蛋白质修饰研究:通过蛋白质组学技术,研究蛋白质的翻译后修饰(如磷酸化、甲基化、乙酰化等),以揭示蛋白质调控的分子机制。
5. 蛋白质组学在疾病诊断和治疗方面的应用:通过蛋白质组学技术,筛选出与疾病相关的蛋白质标志物,为疾病的早期诊断、疾病分类和疾病治疗提供依据。
这些都是生物信息学早期蛋白质研究方面的部分重要工作

7 生物信息学开创者和目标




生物信息学的主要研究目标是利用计算机技术和生物学知识来解决生物学中各种问题,包括但不限于以下方面:
1. 生物序列分析:对基因组、转录组、蛋白质组等生物序列进行分析,以了解其结构、功能和进化关系。
2. 生物系统分析:对生物系统的结构和功能进行建模和分析,以预测其行为和响应外部刺激的能力。
3. 生物计算模拟:利用计算机模拟技术预测生物过程的动态变化和稳态行为,如药物作用机制、代谢途径等。
4. 生物信息学工具开发:开发各类生物信息学工具和软件,如序列比对软件、基因注释软件、通路分析工具等。
5. 生物大数据分析:对生物大数据进行收集、整合和分析,以探索其内在规律和应用价值。
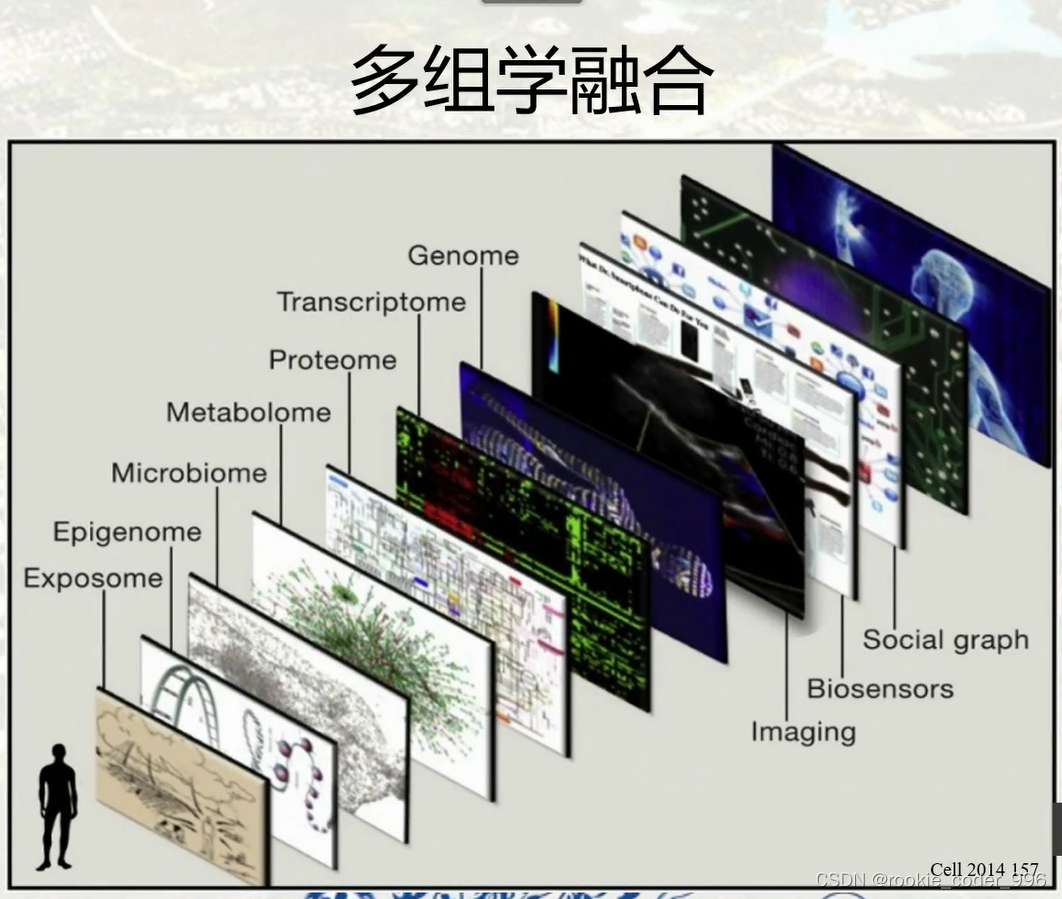
8 生物信息学的研究方向

文章来源:https://uudwc.com/A/DzZEn

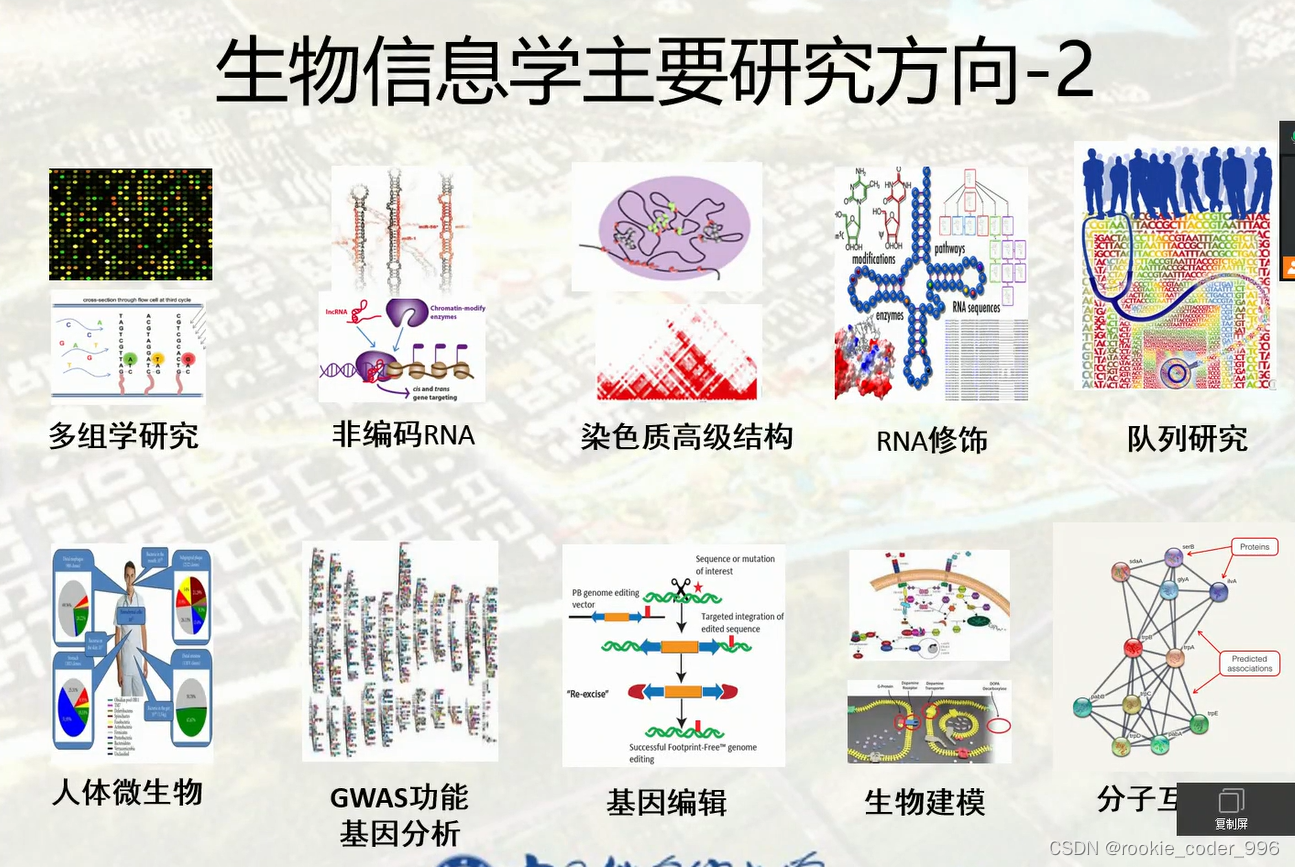
9 蛋白质研究领域的新进展
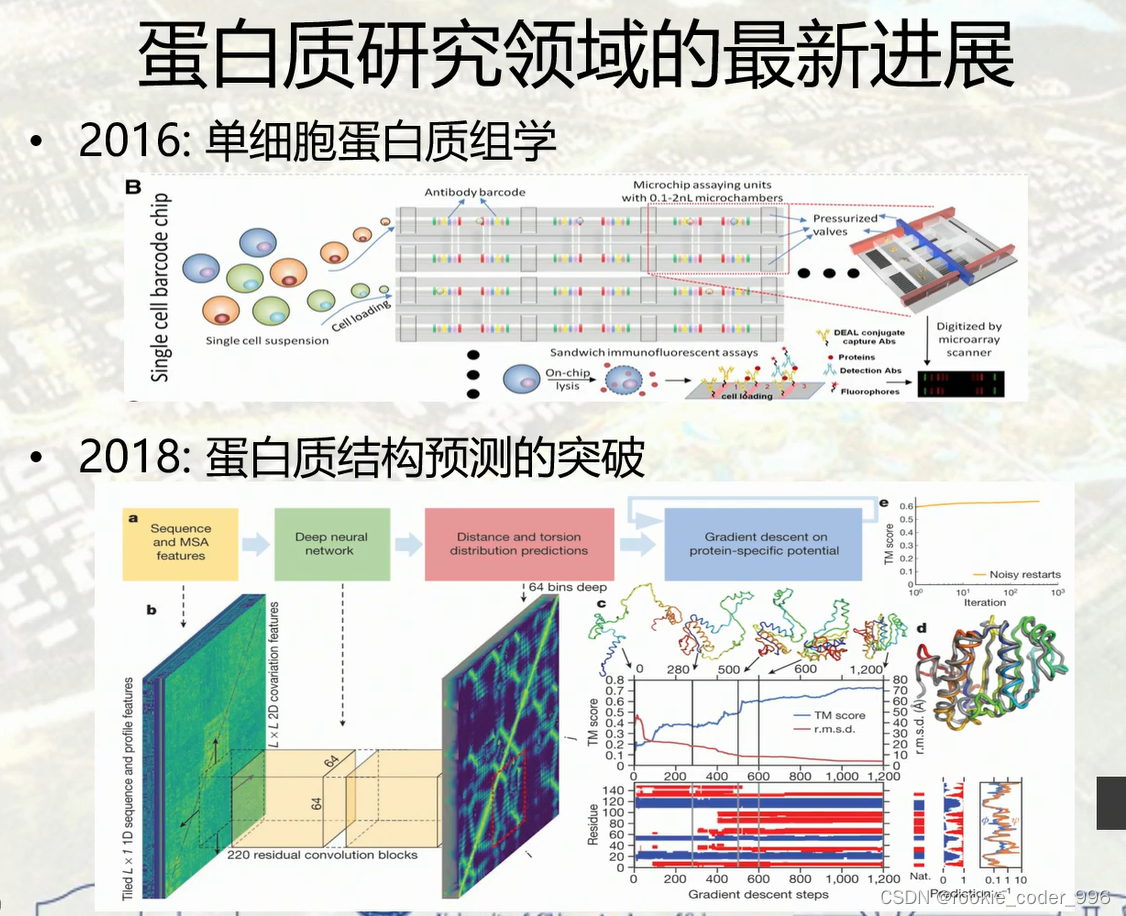
Q:为什么研究单细胞?
A: 为了研究感兴趣的目标细胞类型,专一
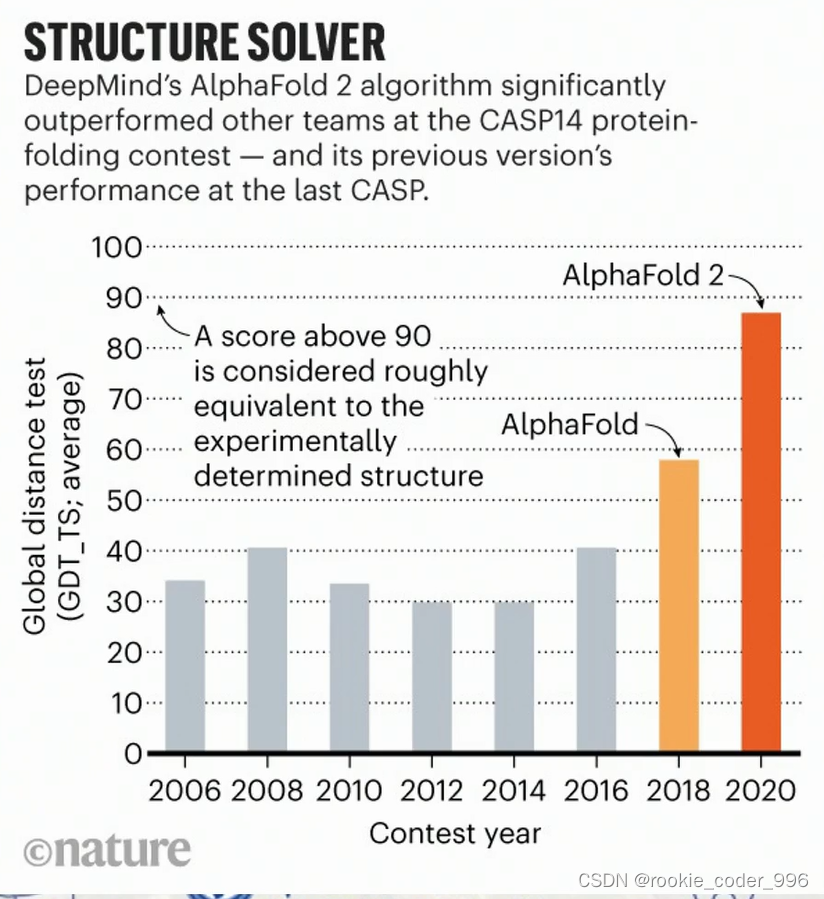
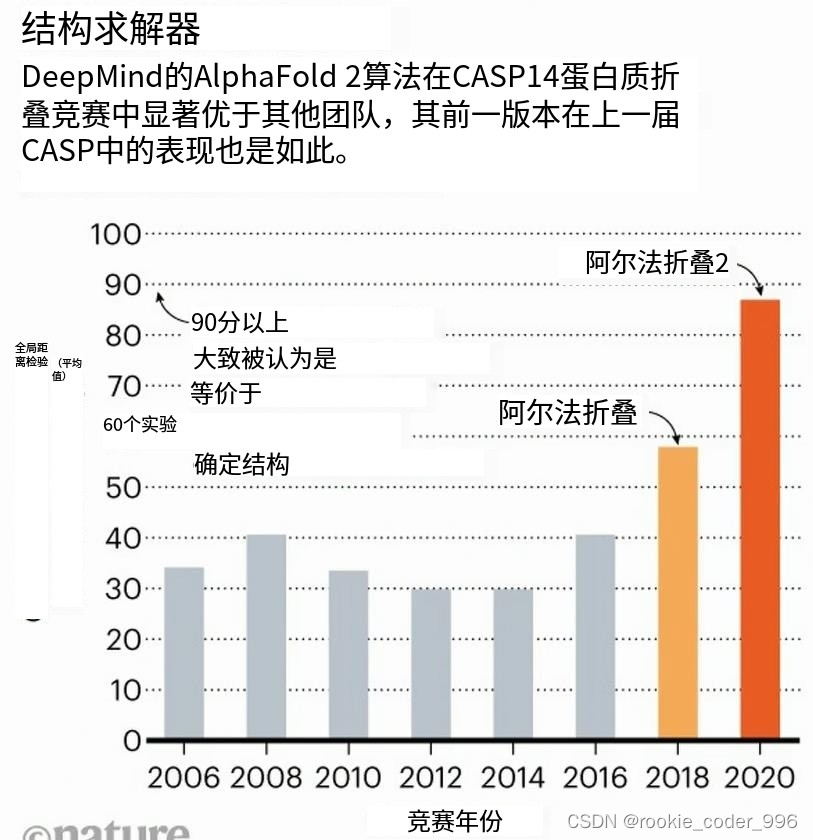
10 AlphaFold
生物信息预测方面做的好一些的公司 是谷歌

DeepMind的AlphaFold是一种人工智能(AI)系统,用于预测蛋白质的三维结构。 AlphaFold使用深度学习技术解决了蛋白质结构预测中的长期挑战,即如何从蛋白质的氨基酸序列推断出它的三维结构。
这个问题被广泛认为是生物学和药物研究领域中最重要的问题之一。
AlphaFold使用神经网络来预测氨基酸序列的蛋白质结构,这使得它比传统的计算方法更准确和更快。在2018年的高管挑战中,AlphaFold获得了最高的分数,并在2021年精确预测了超过200,000个蛋白质结构,这将为开发心血管疾病、癌症和神经退行性疾病的新药物提供帮助。

11 RoseTTAFold

12 基因芯片

- 基因芯片的应用示例
左边是显微镜观察,容易误诊
右边是用基因芯片检测的基因表达情况,对比明显
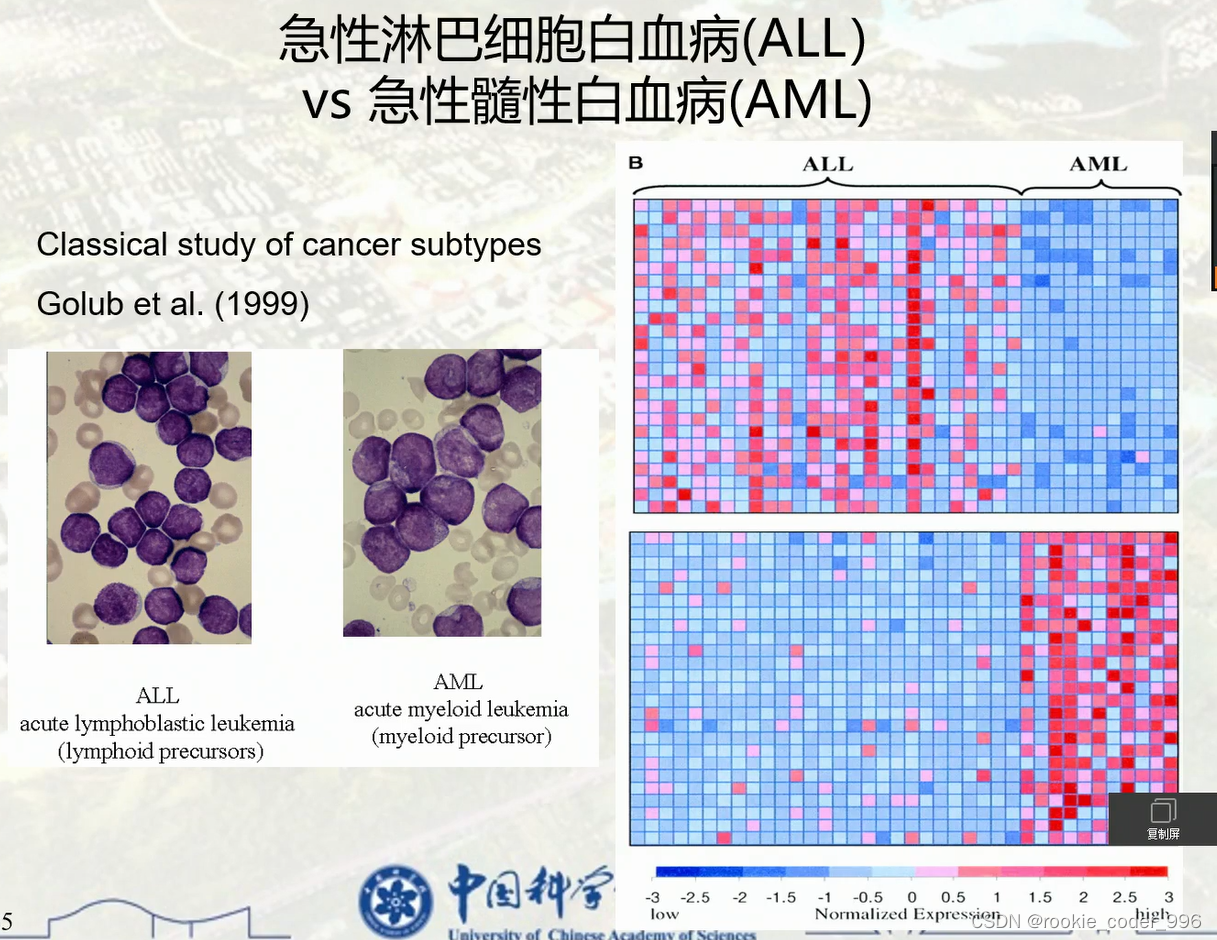
13 基因表达谱芯片
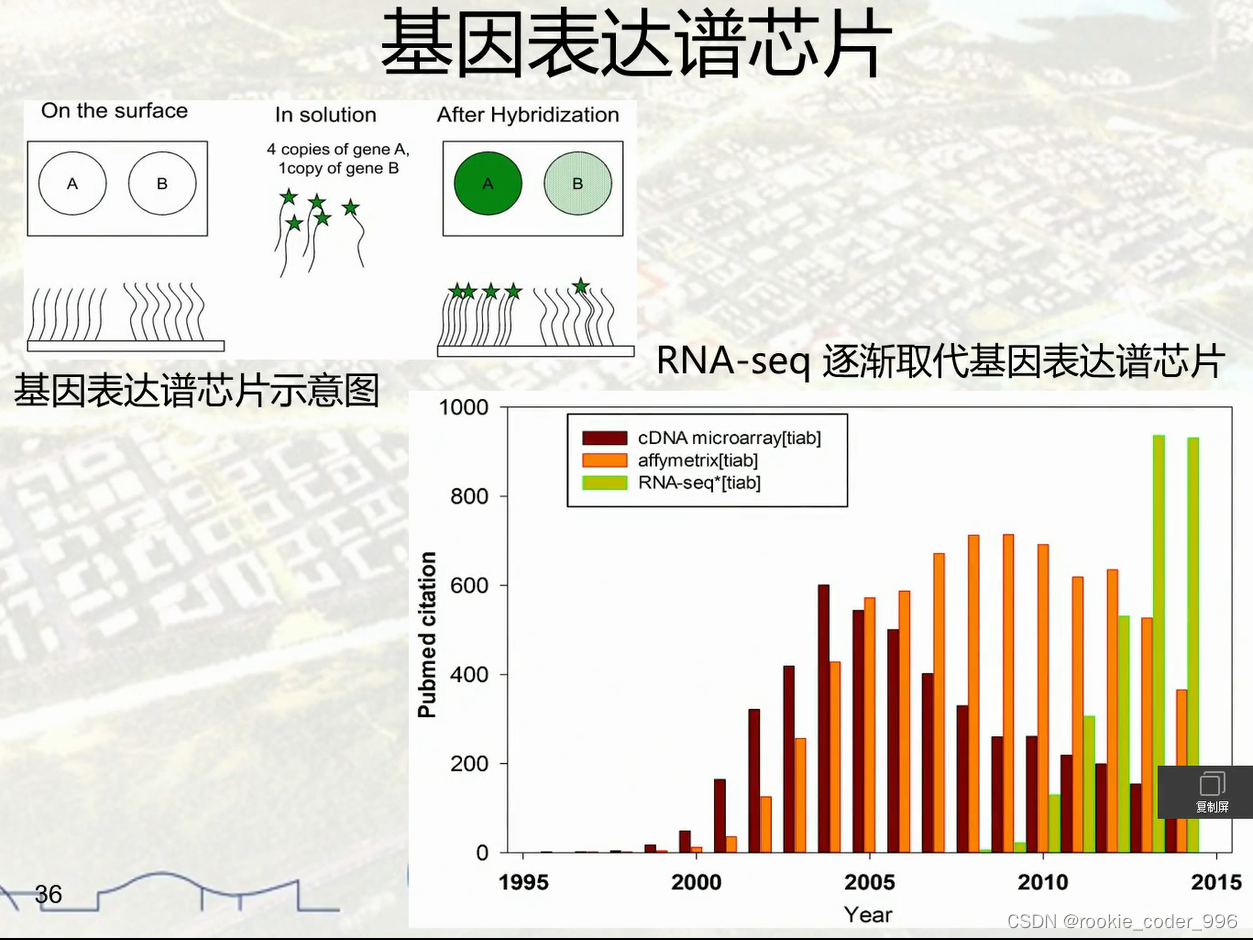
14 SNP芯片
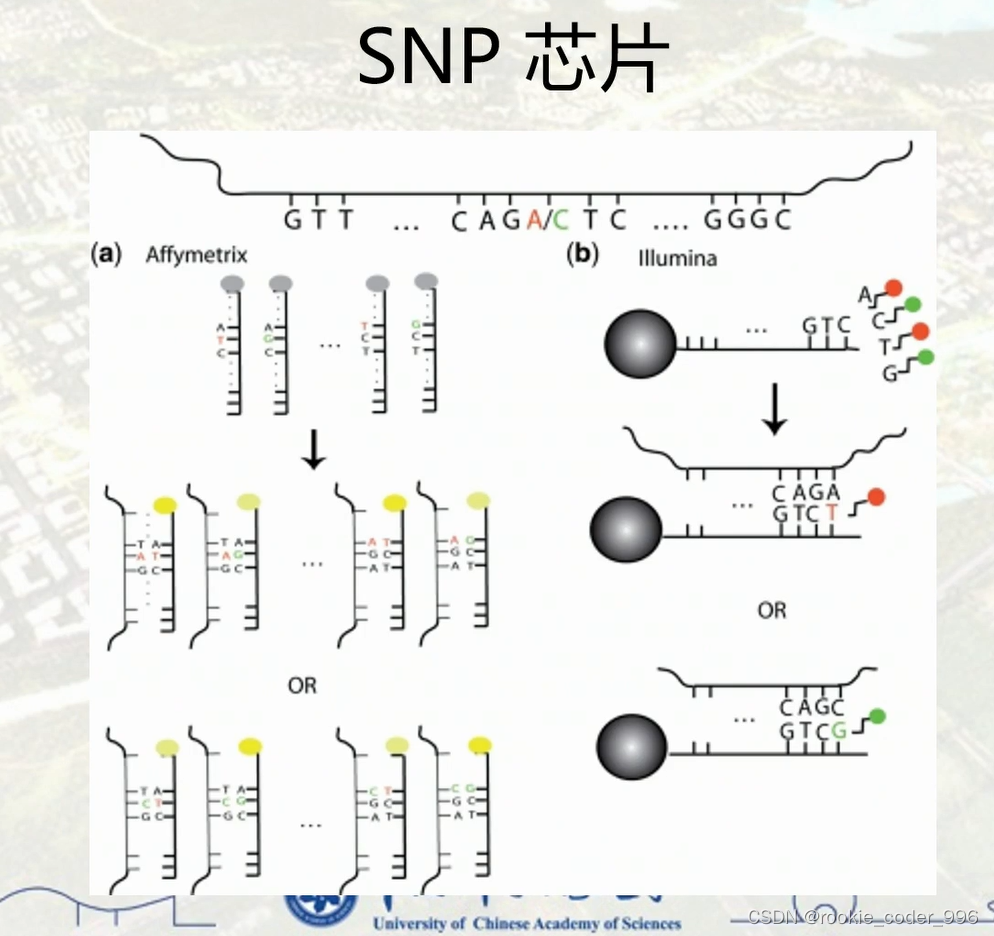
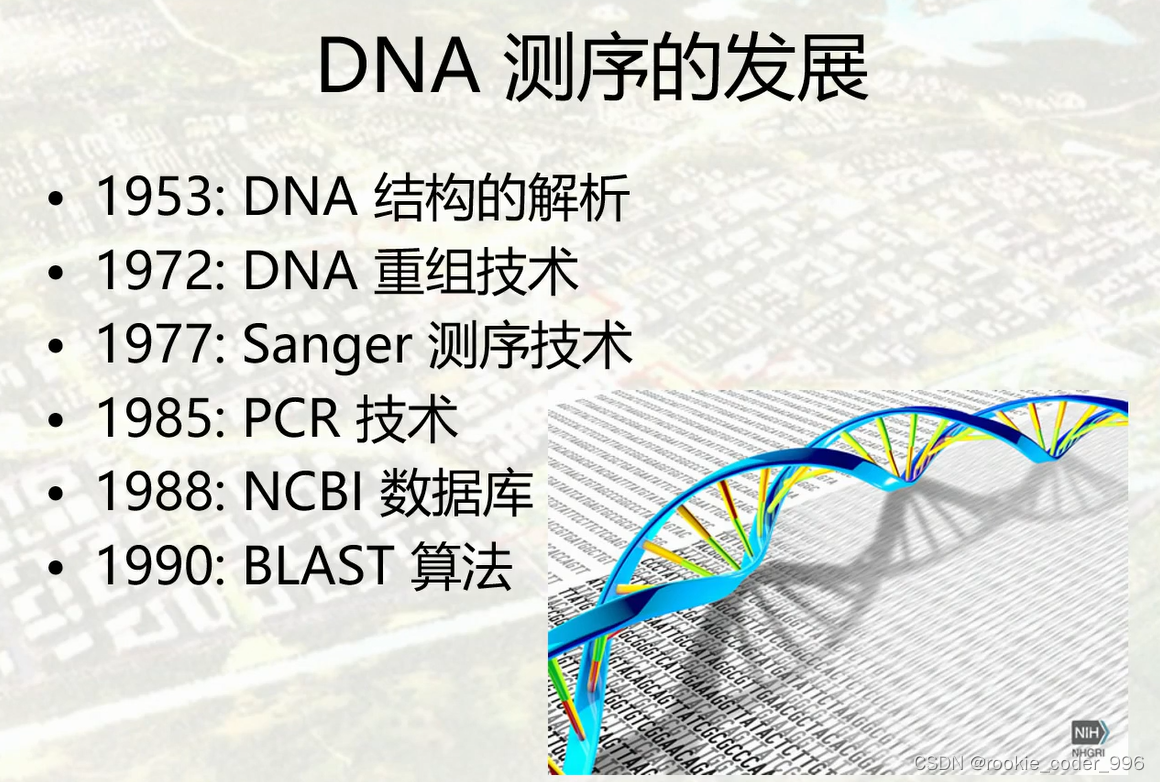
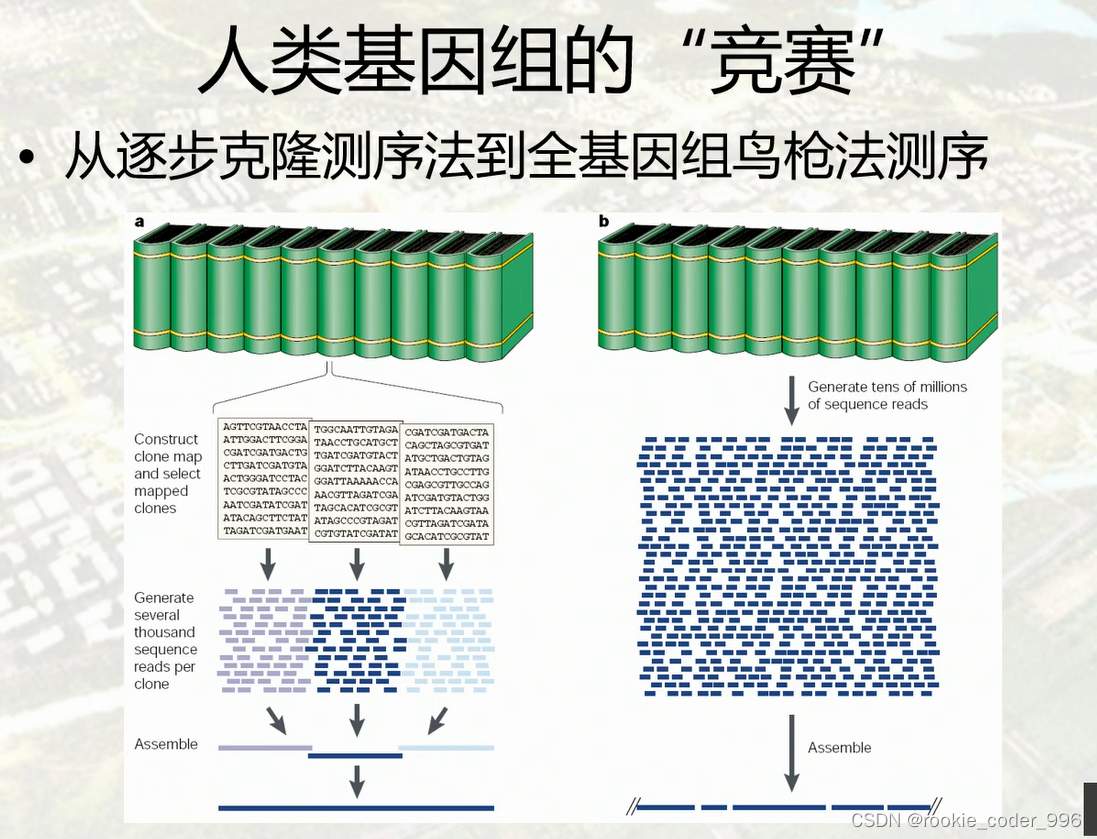
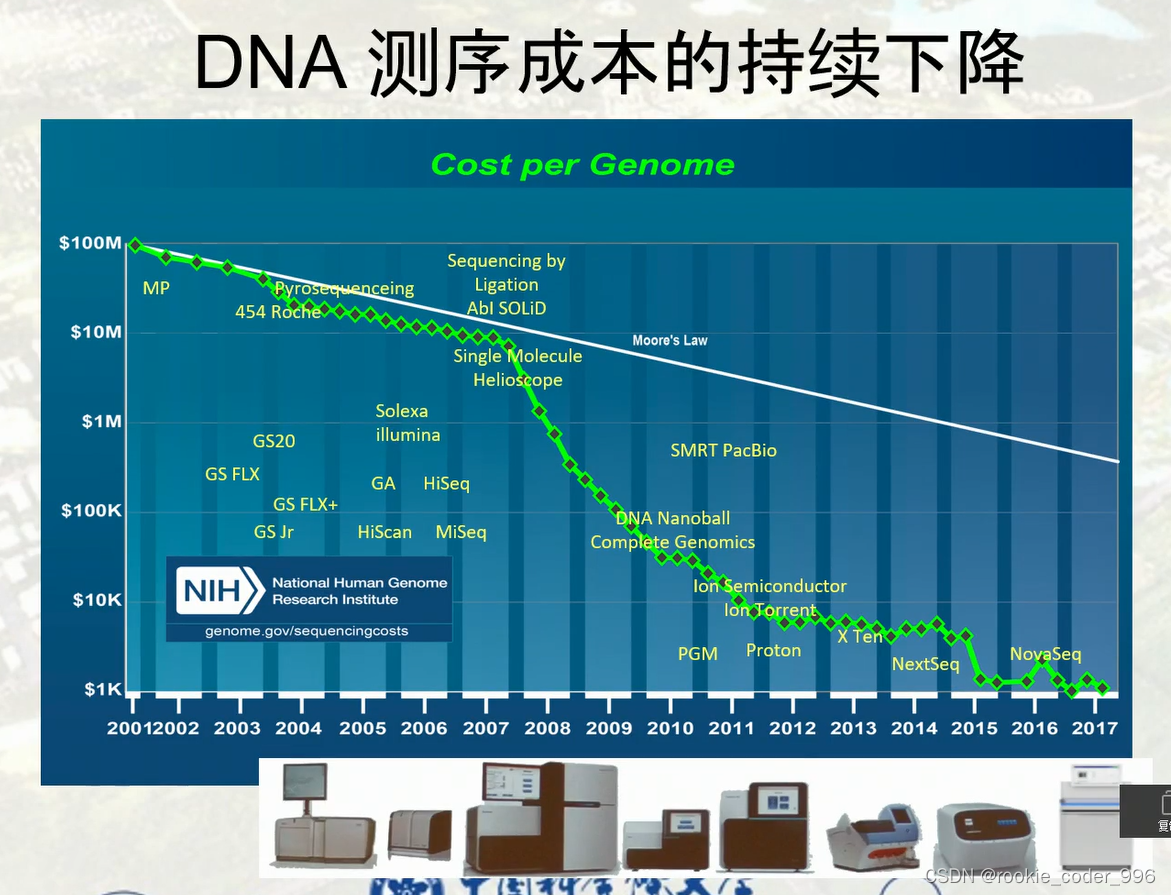
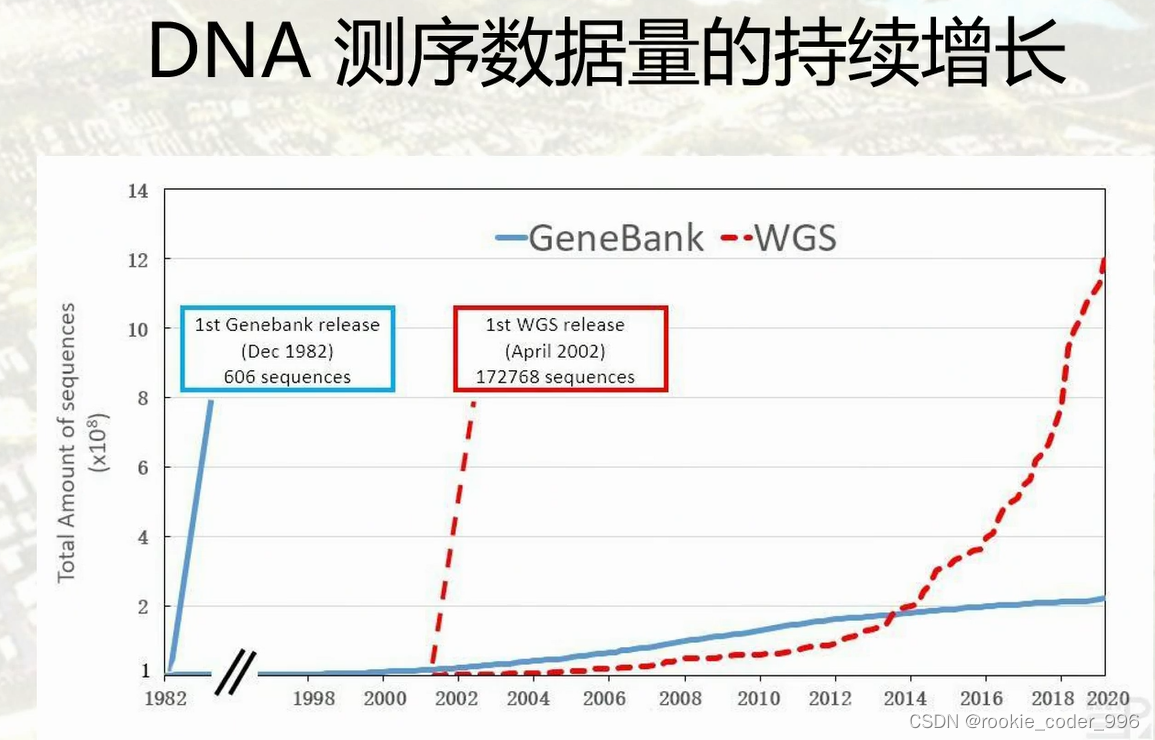
15 DNA测序